그래핀 표면에서의 라카아제 효소의 동적 거동 및 고정: 분자동역학을 이용한 연구
© The Korean Society for Noise and Vibration Engineering
Abstract
The interfacial direct electron transfer (DET) between a biocatalyst and an electrode is crucial for developing electrochemical devices. Graphene is a promising material for high-performance electrodes because of its electric conductivity, material properties, and low cost. However, the interfacial effect of the oxygen reduction enzyme called laccase (LAC) and graphene has not been studied yet. In this study, immobilization of LAC on graphene results in better DET performance compared to that for previous materials. Moreover, molecular dynamics simulation was performed to visualize the interfacial effects, in terms of the binding site, orientation form, and structural affinity. Graphene and LAC were strongly immobilized by vdW force, and the obtained data were validated by comparison with those of other materials. Our research reveals that immobilization of LAC on graphene results in good DET performance through structural changes of graphene.
Keywords:
Electron Transfer, Laccase, Graphene, Molecular Dynamics키워드:
전자전달, 라카아제, 그래핀, 분자동역학1. 서 론
효소 기반 바이오 연료 전지(enzymatic biofuel cells)는 의료 및 상업적 목적으로 30년 이상 사용되어 왔다. 바이오 연료 전지는 미래 에너지 생산을 위한 가장 유망한 재료 중 하나이다. 다른 BFC(biofuel cells)와 달리 EBFC(enzymatic biofuel cells)는 인간의 혈액과 눈물을 사용하여 전자 장치에 전력을 공급할 수 있는 전자를 생성한다. 기질의 산화 환원 효소는 혈액과 눈물의 포도당과 산소와 관련된 화학 반응을 가속화한다. 산화-환원 반응을 촉진하는 단백질 인 효소는 혈당을 이용하여 전기 에너지를 생성한다. 산화(예: 포도당 산화 효소(glucose oxydase) 및 포도당 탈수소 효소(glucose dehydrogenase)) 및 산소 감소(예: 라카아제(laccase) 및 빌리루빈 산화 효소(bilirubin oxydase))를 촉진하는 다양한 효소가 있다. 효율적인 전자 전달(ET)과 효소 촉매 작용을 통해 혈액으로부터 충분한 전력을 저장하기 위해서는 효소가 전극에 강하게 고정되어야 한다(1~4). 효소의 산화 환원 전위는 효소 활성에 중요한 영향을 미친다. 이는 전자 생산 및 전달과 밀접한 관련이 있다(5). 이러한 연료 전지는 웨어러블 장치(6~8)와 같은 소형 전자 장치에 적합할 수 있지만 저전력 출력과 짧은 수명으로 인해 광범위한 응용 분야에 방해가 된다.
EBFC의 성능을 향상시키기 위해 여러 연구가 수행되었으며, 이는 작동 메커니즘에 따라 두 가지 유형, 즉 직접 전자 전달(direct electron transfer, DET) 및 매개 전자 전달(mediated electron transfer, MET) 장치로 분류될 수 있다. 현재, MET는 매개체 사용으로 인해 향상된 산화 환원 전위로 인해 더 높은 전력 출력을 나타낸다. MET의 주요 단점은 복잡한 제조, 단락을 유발하는 구조적 문제 및 비용이다. DET는 전자 전달을 위해 전극과 효소 사이에 불편한 매개체를 필요로 하지 않는다(9). 따라서 DET는 전극 표면에 대한 연구를 통하여 성능 개선의 가능성이 높다. 그럼에도 불구하고 효율적인 DET 과정을 달성하는 데는 몇 가지 근본적인 장애물이 있다. i) 효소가 전극을 향하는 활성 부위의 위치 파악(활성 부위 배향); ii) 효소와 전극 사이의 높은 표면적 달성; 및 iii) 강하게 고정된 효소-전극 복합체를 제작하여 EBFC의 안정성을 향상하는 방법이 있다(8,10~13).
지금까지 Au 및 Au 나노 입자(AuNP)와 같이 높은 전기 전도성을 나타내는 물질은 다양한 표면 변형(예: 공유 결합, 가교 및 자기 조립된 단층)을 가진 EBFC에 사용되어왔다(14,15). 반면 다공성 전극은 고도로 전기 활성이 높은 효소 표면을 생성하여 강화된 ET를 위해 효소 복합체와 효율적인 표면 상호 작용을 수행한다(16). 예를 들어, 탄소 나노 튜브 복합체(예: 단일 벽 및 다중 벽 탄소 나노 튜브(SWCNT 및 MWCNT))는 강한 소수성 상호 작용으로 인해 LAC의 흡수이 뛰어나다(17,18). 최근 유기 용매의 추가 LAC는 탄소 나노 튜브의 배향을 향상시켜 더 큰 전류 밀도를 달성할 수 있게 한다(13). 그러나 EBFC에서 그래핀 기반 전극을 적용하는 것과 관련된 문헌은 매우 부족하다. 그래핀 시트는 EBFC 적용시 높은 투명성과 높은 전기 전도도 및 우수한 물성을 나타내는 2차원 탄소 소재로 일반 흑연에서 파생되어 CNT에 비해 제작이 용이하여 일반적으로 저렴하다. 실제로 그래핀의 표면 특성은 화학적 도핑과 유도체화를 통해 무한히 변형되어 그래핀 산화물과 같은 동족체를 생성할 수 있다(20). Tkac 등(22)은 그래핀 기반의 EBFC와 기타 관련 합성물의 성능을 비교하는 리뷰 기사를 발표했다. 그들은 그래핀이 다른 재료에 비해 더 높은 전력 밀도로 인해 유리하다는 결론을 내렸다.
이 연구의 목적은 그래핀 기반 EBFC의 차별성을 확인하고 성능 향상을 위한 기본 지식을 얻는 것이다. Marcus 이론을 적용하여 서로 다른 효소-전극 계면에서의 ET 상수와 속도를 도출하였다. 또한 그래핀 계면에서 LAC 및 BOD(bilirubin oxidase)와 같은 다중 구리 효소를 수용하는 전자의 고정화를 다루는 문헌은 매우 부족한 실정이다. 따라서 그래핀에 효소를 고정하고 흡수 메커니즘과 원자 규모에서 시뮬레이션을 이용하여 효소와 전극 계면의 구조적 특징을 밝히는 것은 굉장히 중요하다. 분자 동역학(MD) 시뮬레이션은 나노 단위에서 시각화를 가능하게 하고 계면 거동에 대한 정보를 제공한다(23,24,65). 기존에 Au, SWCNT 및 자기 조립 단층(self-assembled monolayer)과 같은 다른 전극 후보를 사용하여 계면 MD 시뮬레이션을 수행한 여러 연구가 있다(25~27). 그럼에도 불구하고 그래핀과의 상호 작용은 아직 밝혀지지 않았다. 현재 시뮬레이션 결과는 그래핀 시트의 효소 고정 메커니즘과 원자 규모의 흡수 거동에 대한 통찰력을 제공하며, 체계적인 실험 검증에 의해 확인되었다. 이 논문은 흡수 및 고정화를 위한 그래핀 기반 전극과 LAC의 여러 변형의 바이오 촉매로서의 가능성을 확인하였다.
2. 재료 및 방법
2.1 분자 모델링: LAC와 그래핀
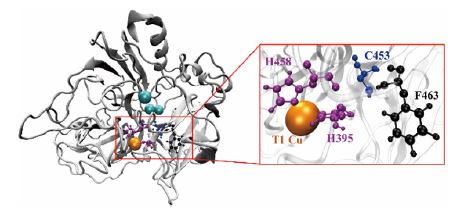
Protein data bank의 구리 함유 산화 효소 LAC, “1GYC”가 야생형으로 선택되었다. 1GYC는 구리 이온의 완전한 구조를 포함하는 산화된 형태의 실온에서 결정으로 추출되었다(28). 누락된 수소 원자는 GROMCAS 5.0.7 소프트웨어를 사용하여 미리 추가되었다(36). MD 시뮬레이션 동안 T1 사이트는 산화된 상태였다. Trametes versicolor의 1GYC는 지금까지 효소 활성에 대한 돌연변이의 영향을 연구하는 데 사용되었기 때문에 선택되었으며, BFC 응용 분야에 유리한 다른 LAC에 비해 더 높은 산화 환원 전위(600 mV ~ 700 mV)를 제공한다(34,37). 그래핀 전극에 고정화 메커니즘을 탐구하기 위해 전극은 LAC 단량체에 맞는 6 nm × 6 nm 및 8 nm × 8 nm 그래핀 시트로 구성되었다(39~41). 그래핀 시트는 VMD의 nanotube builder를 사용하여 구성되었으며 그래핀의 모든 분자역학 매개변수는 automated topology builder(ATB) 웹서버를 사용하여 생성되었다(38).
2.2 분자 동역학 시뮬레이션
세 가지 시뮬레이션 단계의 세부 기능과 목표는 결과 및 토론 섹션에 설명되어 있다. 시뮬레이션을 위한 프로토콜은 다음과 같다. LAC-그래핀 계면에서의 상호 작용은 GROMOS54A7 force-field를 사용하여 GROMACS 5.0.7 소프트웨어로 계산되었다. 경계조건은 십이면체 상자가 있는 시스템에서 1.2 nm의 거리를 두어 설정했다. 그 후, SPC(simple point charge) 물 분자를 십이면체 상자에 추가하고 구리 이온의 1 nm 이내의 물 분자를 제거하여 중첩을 방지했다. 염 농도를 0.15 mol L-1로 설정하고 반대 이온(Na + 및 Cl-)을 첨가하여 시스템을 중화시켰다. 에너지 최소화 과정(energy minimization)은 효소안에 포함된 구리 이온을 고정하고 물이 자유롭게 움직일 수 있도록 허용 오차 1 kJ·mol-1·nm-1에서 가장 가파른 하강 방법을 통해 최대 10 000회 수행되었다. 등온-등온(NVT) 앙상블을 사용한 100 ps MD 시뮬레이션으로 평형을 달성한 다음 각각 1 bar 및 300 K에서 100 ps 등온-등압(NPT) 앙상블을 사용했다. 장거리 정전기 상호 작용은 particle mesh Ewald 방법을 사용하여 계산되었다. 단거리 및 vdW(van der Waals) 상호 작용은 거리 컷오프가 12 Ǻ로 계산되었다. 마지막으로, 안정화 절차 후 2단계에 100 ns, 1단계와 3단계에 50 ns를 적용하여 아무런 제약없이 평형 MD를 수행했다. 모든 궤적은 각 시간 단계에 대해 1 ps로 저장되었다.
2.3 계산 방법을 위한 데이터 분석
LAC의 구조적 안정성을 정량적으로 평가하기 위해 RMSD, 그래핀의 접촉 원자 수, LAC의 회전 반경, LAC와 그래핀 사이의 최소 거리를 계산했다. 고정화 상태를 평가하기 위해 LAC와 그래핀 시트 사이의 거리가 6 Å 미만인 접촉 원자의 수와 최소 거리를 도출하였다. 모든 데이터는 GROMACS 5.0.7 플러그인을 사용하여 계산되었다(36). 각 효소 및 잔기의 접촉 확률은 1 ns 간격으로 전체 시뮬레이션 궤적을 통해 거리를 계산하여 MATLAB 2018a를 이용하여 도출되었다.
결합 친화도를 적절하게 평가하기 위해 각 실행의 마지막 1 ns에 대해 분자역학/푸아송-볼츠만 표면적(MM/PBSA) 분석을 수행하여 LAC-그래핀 계면의 자유 에너지를 계산했다. MM/PBSA는 정확한 사용이 검증되었으며 단백질-리간드 결합, 단백질-단백질 결합 및 생체 분자 상호 작용에 널리 적용된다(43~46). 자유 에너지는 결합 친화도와 상관관계가 있으며 GROMACS의 g_mmpbsa 모듈을 사용하여 계산되었다(47).
3. 결과 및 토론
3.1 분자 동역학을 통한 Laccase-graphene 계면 평가
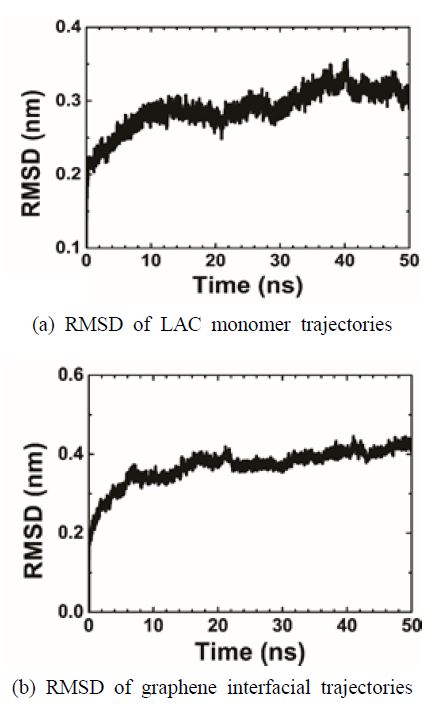
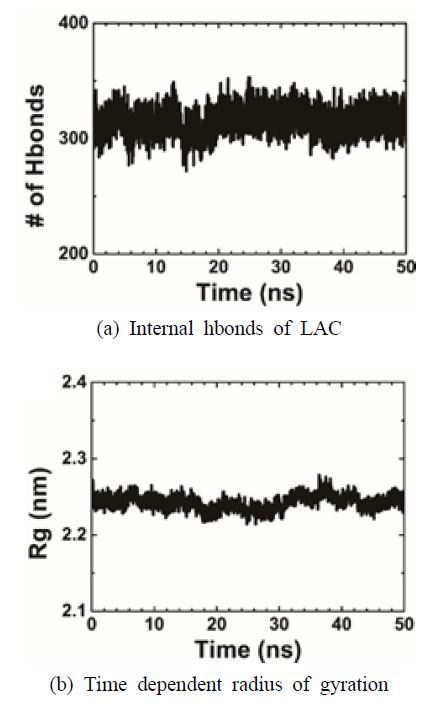
그래핀에 효소 모델을 정렬하기 전에 각 단량체에 대해 평형 분자동역학 시뮬레이션을 수행하였다. 이것은 1단계로 정의한다. MD 시뮬레이션은 효소 단량체에 대해 50 ns에 대해 수행되었으며 총 시뮬레이션 시간은 150 ns이다(Fig. 1 참조).
각 모델의 궤적과 평형을 연구하기 위해 용액에 4개의 구리 이온을 포함하는 LAC의 root mean square deviation(RMSD)을 계산했다. Fig. 2는 4개의 구리 이온을 포함하는 효소에 대한 1단계의 최종 형태를 보여준다. Fig. 2는 RMSD가 40 ns의 분자 역학(MD) 궤적에 따라 0.3 nm의 값에서 포화됨을 보여준다. 40 ns의 MD 궤적에 따른 평균 및 표준편차값은 0.315±0.01이다. RMSD 값은 평형 상태에 도달하여 이 데이터를 통하여 각 모델의 단량체가 안정화되었고 그래핀에 고정화를 위한 평형 구조에 도달했음을 알 수 있다.
그래핀에 LAC가 결합하는 메커니즘을 이해하기 위에 효소 크기를 고려하여 6 nm×6 nm의 그래핀 시트를 제작했다. 이 시뮬레이션은 2단계로 정의된다. LAC-그래핀 계면은 LAC-CNT 계면과 유사한 소수성 상호 작용에 의해 대부분 영향을 받는 것으로 예측된다(25,61). 효소 표면에서 정보를 수집합니다.
Fig. 3은 시간 종속 시뮬레이션에서 얻은 초기에서 최종 궤적을 보여준다. 5 ns 후, 두 개의 그래핀 시트가 T1 Cu 사이트에 쌓였다. 또한 다른 면에 위치했던 그래핀은 10 ns의 시뮬레이션 후 T1 Cu 사이트에서 세 번째 층으로 적층 되었다. 10 ns 시뮬레이션 후 최대 100 ns까지 동일한 모양이 유지되었으며, 동적 거동을 통하여 최종 구조를 확인할 수 있다. 바닥의 3층 적층 그래핀 시트와 LAC의 각 면에 있는 2개의 그래핀 시트도 지속적으로 유지되었다.
다음으로 결합 시뮬레이션에서 가장 결합 빈도가 높은 아미노산을 확인하여 특징 분석을 하였다. 가장 높은 빈도를 나타낸 아미노산은 LYS194, PHE265, THR292, PHE332, ALA362 이며, 잔기 중 4개는 소수성 특징을 가지고 있다(63). 또한, 3층 적층 그래핀 시트는 T1 Cu 사이트에 매우 밀접하게 결합되어 있다. 따라서 분자동역학 시뮬레이션을 통하여 우리는 다음 두 가지 특징을 발견하였다. i) 결합 잔기는 소수성이며 이는 효소-그래핀 계면에 매우 중요하다. ii) 그래핀 시트에는 LAC에 대한 세 가지 주요 잠재적 결합 부위가 있다. 특히, T1 Cu 사이트에 가까운 기판 입구 근처의 사이트는 가장 강한 결합 사이트이며 DET를 개선하기 위한 배향 목표가 될 수 있다.
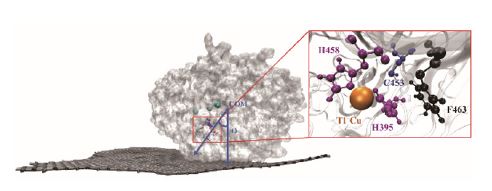
고정화 과정, 활성 부위의 배향 특징, 결합 형태, 결합 친화도 및 DET를 조사하기 위해 그래핀과 LAC에 고정을 분석하기 위하여 50 ns MD 시뮬레이션을 수행했다. 이것은 3단계로 정의된다. 효소는 Fig. 4와 같이 8 nm × 8 nm 그래핀 시트에 고정화되었다. 효소의 초기 위치는 그래핀 시트의 방향을 고려하여 12 Å 거리에서 T1 Cu 결합 부위를 그래핀 시트를 향해 정렬되었다. MD 궤적은 SEM 이미지에 의해 그래핀 전극에서 1GYC에 대해 확인된 바와 같이, 그래핀 시트에 잘 고정되어 있음을 보여준다. 2D 평면에서 효소-전극 계면의 특징을 이해하기 위해 3단계에서 그래핀 시트의 크기를 2단계보다 2 nm 더 넓게 설정했다. 가장 접촉 빈도가 높은 아미노산을 분석해보니 1단계에서 관찰된 경향과 유사하게 16개 잔기 중 12개는 소수성 잔기를 포함하는 아미노산이였다.
효소의 흡수도는 중심에서 T1 부위 벡터와 중심에서 그래핀 벡터 사이의 각도를 계산하여 평가했다. 각도는 50 ns MD 궤적의 마지막 5 ns를 추출한 다음 평균을 구하여 계산하였다. 그래핀 시트는 MD 시뮬레이션의 X-Y 전역 좌표에 배치되었다. Fig. 4는 전극의 표면 흡수도와 각도 θ를 41.952°임을 보여준다.
Fig. 4는 2단계에서 얻은 결과와 같이 T1 Cu 부위가 특정 결합 부위로 적절하게 배향되어 있음을 보여준다. 따라서 더욱 많은 표면이 그래핀에 고정되고 T1 Cu 부위와 그래핀 사이의 거리가 가까움을 확인하였다.
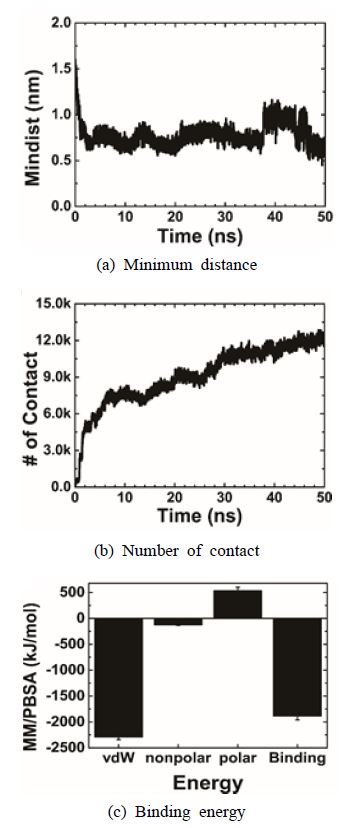
LAC가 전체 시뮬레이션 궤적에서 확실하게 고정되었는지 확인하기 위해 효소와 그래핀 사이의 최소 거리를 계산하였다. Fig. 5(a)에서 볼 수 있듯이 최소 거리는 3 ns ~ 9 ns 후에 0.74±0.028 nm에서 수렴되었다. 접촉 횟수의 경우 효소와 그래핀 사이의 거리가 0.6 nm 미만을 기준으로 계산하였다. 따라서 MD는 3개의 LAC가 전체 궤적에 걸쳐 그래핀에 적절하게 흡수되었음을 보여준다.
흡수된 효소의 동적 및 구조적 특징은 이전 연구에서 수행된 계산 값과 비교하며 분석하였다(39~41). Figs. 5 ~ 6을 확인하면, 2개의 LAC-그래핀 시스템에 대한 RMSD값은 23 ns 후 0.39 nm에서 0.52 nm 사이에서 포화되었다. 수소결합의 평균 수는 WT 및 F463Y에 대해 각각 315±10 및 327±16이었다. 회전 반경은 50 ns MD 궤적의 평균에 대해 2.24±0.009 nm 및 2.24±0.016 nm였다. 시뮬레이션을 통하여 시간에 따른 데이터를 확인함으로써 수소 결합 및 회전 반경 결과는 그래핀에 흡수된 효소가 MD 궤도에 걸쳐 매우 안정적인 구조를 유지함을 보여준다.
그래핀 계면에서 LAC의 구조적 변화를 연구하기 위해 먼저 1단계(벌크 솔루션)와 3단계(그래 핀에 고정됨)의 평균 RMSD 차이를 비교했다. 여기에서 거리는 0.080 nm 및 0.083 nm임을 확인했다. 이 결과는 Zho 등이 수행한 다른 분자 연구에서 얻은 결과와 상당히 비교됨을 알 수 있다. 양전하 또는 음전하 SAM에 흡수된 LAC의 경우 총 RMSD가 크게 변하지 않는 것으로 나타났다(0.05 nm 미만)(26). 그러나 그래핀 계면에서 흡수된 정도는 SAM 계면에서 보다 최대 3배나 더 크다. 이것은 결합 특징이 효소-기질 계면에서 물질의 특성에 의해 지배된다는 것을 의미하며, 더 강한 결합은 고정화 효능의 긍정적인 지표이다. 양전하 또는 음전하 SAM은 효소 구조를 유지하면서 동시에 LAC의 방향을 변경할 수 있지만 그래핀에 비해 흡수 강도가 좋지 않다. 또한, LAC는 MD 시뮬레이션을 기반으로 고정된 표면보다는 그래 핀에 흡수된 후 더 많은 형태적 변화를 겪는 것으로 밝혀졌다. 이 연구에서의 Au와 그래핀의 전류 밀도를 비교한 데이터는 이 주장을 뒷받침하며 그래핀에서의 고정력은 우수하고 구조적 안정도가 높음을 확인하였다.
또한, LAC는 MD 시뮬레이션을 기반으로 고정된 표면보다는 그래 핀에 흡수된 후 더 많은 형태적 변화를 겪는 것으로 밝혀졌다. 이 연구에서의 Au와 그래핀의 전류 밀도를 비교한 데이터는 이 주장을 뒷받침하며 그래핀에서의 고정력은 우수하고 구조적 안정도가 높음을 확인하였다.
접촉 원자의 수와 결합 에너지를 측정하여 결합 친화도를 정량적으로 비교할 수 있다. Fig. 5(b)는 1GYC의 경우 최대 12 226으로 전체 원자 개수에 비해 34 % 정도의 원자가 접촉하는 것을 확인하였다. Fig. 5(c)에 제시된 결합 에너지 데이터는 비 결합 상호 작용, 특히 vdW 힘이 총 결합 에너지를 결정하는 데 중요한 역할을 한다는 것을 보여준다. 순수한 그래핀 시트는 순 전하가 0이기 때문에 정전기 상호 작용을 겪지 않기 때문에 분자동역학을 사용하여 계산된 에너지에는 정전기 에너지가 포함되지 않았다. 결합 에너지는 1888 kJmol-1로 vdW, nonpolar 그리고 polar 에너지의 총합을 바탕으로 계산되었다. MD 데이터의 종합하면 LAC-그래핀 계면에서의 결합 친화도도 축 리간드의 소수성에 의해 영향을 받는다는 것을 알 수 있다.
3.2. Marcus 이론 및 전자 커플 링에 의한 전자 전달 역학 평가
전자전달 이론을 정립한 Marcus 이론에 따르면 효소와 그래핀 사이의 계면 DET는 T1 Cu 사이트가 전극 표면에 충분히 가까운 경우에만 발생할 수 있다. 용액에서 약하게 결합된 전자 공여체 A와 수용체 B 사이의 전자 전달 속도 상수는 식 (1)에 의해 설명된다.
| (1) |
방정식의 상수는 h (플랑크 상수), R (이상 기체 상수) 및 KB (볼츠만 상수)이다. HAB는 A와 B 사이의 전자 커플링, T 는 온도, ΔG는 ET의 자유에너지 차이, λ는 재구성 에너지이다. 여기서 전자 커플링은 주로 다음 수식의 HAB에 의해 결정된다.
| (2) |
여기서 HAB0은 vdW 거리(r0)에서의 전자 결합입니다. 터널 매개변수 β는 단백질에 대한 ET의 효율성을 반영하며 단백질 구조에 따라 다르다. 따라서 이 이론에 따르면 계면 DET에 영향을 미치는 가장 중요한 요소 중 하나는 활성 부위와 전극 표면 사이의 거리가 된다(62). 계면 ET 속도의 거리 의존성은 효소-전극 어셈블리 뿐만 아니라 다양한 생체 분자(예: DNA, 펩타이드 및 단백질)의 ET에서도 관찰된다(63,64). Crespilho 등(65)은 GOx와 전극 표면을 포함하는 산화 환원 중심에 대한 ET 속도를 거리에 따라서 분석하였다. 그들은 서로 다른 전극 재료에 대해 동일한 β를 가정하여 전자 커플 링을 단순화하고 다양한 생성된 효소 전극 모델의 ET 비율에 대한 비율을 제안했다. 따라서 MD 연구를 통하면 T1 Cu 사이트의 중심 좌표와 그래핀 사이의 최소 거리를 바탕으로 ET 속도 상수를 계산할 수 있다.
MD 시뮬레이션에서 결정된 평균 d는 0.78±0.12였으며, 이는 전자 커플링의 최소 단위인 ~ 1.4 nm의 거리 범주 안에 포함됨을 확인하였다. 최종적으로, ET 비율의 비교는 식 (1)과 (2)에서 β값이 동일하다고 가정한다면 식 (3)과 같이 나타낼 수 있다.
| (3) |
한편으론, MD는 원자 규모에서 단량체 효소와 그래핀의 단층 사이의 계면을 고려하는 반면 습식 실험에서는 효소 응집, 배향 및 전극의 고정되지 않은 효소를 고려해야하기에 실험과 시뮬레이션의 통합은 전극 표면 개발을 효율적으로 하는 방향성을 제시한다.
더 나아가 MD 시뮬레이션은 고정화 프로세스에 대한 자세한 통찰력을 제공하고 ET 성능을 향상시키는 매개 변수를 강조하여 향후 효소 개량의 가능성을 보여준다. 한편으론 야생형 LAC는 강력한 vdW 및 소수성 상호 작용을 통해 T1 Cu 사이트를 통해 그래핀에 결합한다는 결론을 내릴 수 있다. 이것은 야생형 LAC도 효소가 추출되는 종에 따라 다양한 특성을 가지기에 이 연구에서 사용된 “1GYC”는 그래핀 전극에 적용하기에 적합하다는 점을 알려준다. 또한, 축 리간드의 소수성은 효소-전극 계면인 외부 측면의 거동을 변화시키기 때문에 효소 내부 부위의 산화 환원 전위 뿐만 아니라 결합 친화도에 영향을 미친다. 따라서 효소 활성 부위와 그래 핀 사이의 거리에 따라 결합 친화도와 ET 속도 데이터를 변화를 바탕으로 효소 개량을 통하여 바이오 전극의 효율을 개선할 수 있다는 점을 시사한다. 최종적으로 LAC-그래핀 계면에 대한 이 연구는 EBFC에 대한 그래핀의 활용도를 확장할 것으로 기대한다.
4. 결 론
이 연구에서는 LAC 효소와 그래핀 전극 사이에서의 계면 특성을 분자동역학 계산 방법을 통하여 분석하였다. LAC-그래핀 계면의 MD 연구는 원자 규모의 효소 고정화 과정을 이해하고 DET 성능을 향상시키기 위해 어떤 매개 변수를 조작할 수 있는지 밝히기 위해 수행되었다. 첫째, 다중 그래핀-LAC 시뮬레이션에서는 향상된 DET를 달성하기 위해 전자 수용체 역할을 하는 T1 Cu 부위에 가까운 특정 결합 부위의 방향을 고려해야 함을 보여주었다. 둘째, LAC를 그래핀에 고정하여 흡수하는 과정에서 결합 형태와 ET 속도를 확인하였다. MD 결과는 축 리간드의 소수성이 활성 부위의 내부 좌표에 약간만 영향을 주지만 결합 친화도 및 ET 속도에도 영향을 미친다는 것을 보여준다. 또한, 우리는 MD를 사용한 구조적 형태 비교를 통하여 그래핀에 고정되었을 때 기존 전극에 비하여 LAC가 더 많이 흡수되는 것을 관찰하였다. 결론적으로, 시뮬레이션 방법을 통하여 실험에서는 볼 수 없는 LAC-그래핀 계면에서의 동적 특성을 분석함으로써 향상된 ET 속도 및 고정화를 가능함을 입증하였다. 더 나아가 이 MD 결과는 효소 개량 부분을 실험적으로 검증하기에 앞서 시뮬레이션으로 예측하여 비용을 저감하고 바이오 전극의 성능을 개선할 수 있는 가능성을 보여주었다.
기 호 설 명
| h : | planck constant |
| R : | gas constant |
| KB : | boltzmann constant |
| HAB : | electric coupling between A and B |
| T : | temperature in Kelvin |
| ΔG : | free energy difference |
| λ : | reorganization energy |
Acknowledgments
이 논문은 2020년도 추계 소음진동 학술대회에서 학생 포스터 발표 우수 논문상을 받은 연구이자 2021년도 정부의 재원으로 한국연구재단의 지원을 받아 수행된 연구임(NRF-2018H1A2A1062291 & NRF-2019R1A2C1086103).
References
-
Fernandez-Fernandez, M., Sanromán, M. Á. and Moldes, D., 2013, Recent Developments and Applications of Immobilized Laccase, Biotechnology Advances, Vol. 31, No. 8, pp. 1808~1825.
[https://doi.org/10.1016/j.biotechadv.2012.02.013]

-
Chung, M., Nguyen, T. L., Tran, T. Q. N. et al., 2018, Ultrarapid Sonochemical Synthesis of Enzyme-incorporated Copper Nanoflowers and Their Application to Mediatorless Glucose Biofuel Cell, Applied Surface Science, Vol. 429, pp. 203~209.
[https://doi.org/10.1016/j.apsusc.2017.06.242]

-
Christwardana, M., Kim, D. H., Chung, Y. et al., 2018, A Hybrid Biocatalyst Consisting of Silver Nanoparticle and Naphthalenethiol Self-assembled Monolayer Prepared for Anchoring Glucose Oxidase and Its Use for an Enzymatic Biofuel Cell, Applied Surface Science, Vol. 429, pp. 180~186.
[https://doi.org/10.1016/j.apsusc.2017.07.023]

-
Castrovilli, M. C., Bolognesi, P., Chiarinelli, J. et al., 2019, The Convergence of Forefront Technologies in the Design of Laccase-based Biosensors – An Update, TrAC Trends in Analytical Chemistry, Vol. 119, 115615.
[https://doi.org/10.1016/j.trac.2019.07.026]

-
Cadet, M., Gounel, S., Stines-Chaumeil, C. et al., 2016, An Enzymatic Glucose/O2 Biofuel Cell Operating in Human Blood, Biosensors and Bioelectronics, Vol. 83, pp. 60~67.
[https://doi.org/10.1016/j.bios.2016.04.016]

-
Tahar, A. B., Romdhane, A., Lalaoui, N. et al., 2018, Carbon Nanotube-based Flexible Biocathode for Enzymatic Biofuel Cells by Spray Coating, Journal of Power Sources, Vol. 408, pp. 1~6.
[https://doi.org/10.1016/j.jpowsour.2018.10.059]

-
Escalona-Villalpando, R. A., Ortiz-Ortega, E., Bocanegra-Ugalde, J. P. et al., 2019, Clean Energy from Human Sweat Using an Enzymatic Patch, Journal of Power Sources, Vol. 412, pp. 496~504.
[https://doi.org/10.1016/j.jpowsour.2018.11.076]

-
Cosnier, S., Gross, A. J., Le Goff, A. et al., 2016, Recent Advances on Enzymatic Glucose/Oxygen and Hydrogen/Oxygen Biofuel Cells: Achievements and Limitations, Journal of Power Sources, Vol. 325, pp. 252~263.
[https://doi.org/10.1016/j.jpowsour.2016.05.133]

-
Bullen, R. A., Arnot, T. C., Lakeman, J. B. et al., 2006, Biofuel Cells and Their Development, Biosensors and Bioelectronics, Vol. 21, No. 11, pp. 2015~2045.
[https://doi.org/10.1016/j.bios.2006.01.030]

-
Mano, N. and de Poulpiquet, A., 2017, O2 Reduction in Enzymatic Biofuel Cells, Chemical Reviews, Vol. 118, No. 5, pp. 2392~2468.
[https://doi.org/10.1021/acs.chemrev.7b00220]

-
Lee, Y. S., Baek, S., Lee, H. et al., 2018, Construction of Uniform Monolayer - and Orientation - Tunable Enzyme Electrode by a Synthetic Glucose Dehydrogenase without Electron-transfer Subunit Via Optimized Site-specific Gold-binding Peptide Capable of Direct Electron Transfer, ACS Applied Materials & Interfaces, Vol. 10, No. 34, pp. 28615~28626.
[https://doi.org/10.1021/acsami.8b08876]

-
Kahoush, M., Behary, N., Cayla, A. et al., 2019, Surface Modification of Carbon Felt by Cold Remote Plasma for Glucose Oxidase Enzyme Immobilization, Applied Surface Science, Vol. 476, pp. 1016~1024.
[https://doi.org/10.1016/j.apsusc.2019.01.155]

-
Wu, F., Su, L., Yu, P. and Mao, L., 2017, Role of Organic Solvents in Immobilizing Fungus Laccase on Single-walled Carbon Nanotubes for Improved Current Response in Direct Bioelectrocatalysis, Journal of the American Chemical Society, Vol. 139, No. 4, pp. 1565~1574.
[https://doi.org/10.1021/jacs.6b11469]

-
Lalaoui, N., Rousselot-Pailley, P., Robert, V. et al., 2016, Direct Electron Transfer between a Site Specific Pyrene-modified Laccase and Carbon Nanotube/Gold Nanoparticle Supramolecular Assemblies for Bioelectrocatalytic Dioxygen Reduction, ACS Catalysis, Vol. 6, No. 3, pp. 1894~1900.
[https://doi.org/10.1021/acscatal.5b02442]

-
Dagys, M., Laurynėnas, A., Ratautas, D. et al., 2017, Oxygen Electroreduction Catalysed by Laccase Wired to Gold Nanoparticles Via the Trinuclear Copper Cluster, Energy & Environmental Science, Vol. 10, No. 2, pp. 498~502.
[https://doi.org/10.1039/C6EE02232D]

-
Holmberg, S., Rodriguez-Delgado, M., Milton, R. D. et al., 2015, Bioelectrochemical Study of Thermostable Pycnoporus Sanguineus CS43 Lacase Bioelectrodes based on Prolytic Carbon Nanofibers for Bioelectrocatalytic O2 Reduction. ACS Catalysis, Vol. 5, No. 12, pp. 7507~7518.
[https://doi.org/10.1021/acscatal.5b01600]

-
Lalaoui, N., David, R., Jamet, H. et al., 2016, Hosting Adamantane in the Substrate Pocket of Laccase: Direct Bioelectrocatalytic Reduction of O2 on Functionalized Carbon Nanotubes, ACS Catalysis, Vol. 6, No. 7, pp. 4259~4264.
[https://doi.org/10.1021/acscatal.6b00797]

-
Tavares, A. P., Silva, C. G., Dražić, G. et al., 2015, Laccase Immobilization over Multi-walled Carbon Nanotubes: Kinetic, Thermodynamic and Stability Studies, Journal of Colloid and Interface Science, Vol. 454, pp. 52~60.
[https://doi.org/10.1016/j.jcis.2015.04.054]

-
Li, D. and Kaner, R. B., 2008, Graphene-based Materials, Science, Vol. 320, No. 5880, pp. 1170~1171.
[https://doi.org/10.1126/science.1158180]

-
Shi, Y., Kim, K. K., Reina, A., Hofmann, M., Li, L. J. and Kong, J., 2010, Work Function Engineering of Graphene Electrode Via Chemical Doping, ACS Nano, Vol. 4, No. 5, pp. 2689~2694.
[https://doi.org/10.1021/nn1005478]

-
Rewatkar, P. and Goel, S., 2019, 3D Printed Bioelectrodes for Enzymatic Biofuel Cell: Simple, Rapid, Optimized and Enhanced Approach, IEEE Transactions on Nanobioscience, Vol. 19, No. 1, pp. 4~10.
[https://doi.org/10.1109/TNB.2019.2941196]

-
Filip, J. and Tkac, J., 2014, Is Graphene Worth Using in Biofuel Cells? Electrochimica Acta, Vol. 136, pp. 340~354.
[https://doi.org/10.1016/j.electacta.2014.05.119]

-
Zhao, D., Li, L., He, D. and Zhou, J., 2016, Molecular Dynamics Simulations of Conformation Changes of HIV-1 Regulatory Protein on Graphene, Applied Surface Science, Vol. 377, pp. 324~334.
[https://doi.org/10.1016/j.apsusc.2016.03.177]

-
Zhao, D., Li, L. and Zhou, J., 2018, Simulation Insight into the Cytochrome C Adsorption on Graphene and Graphene Oxide Surfaces, Applied Surface Science, Vol. 428, pp. 825~834.
[https://doi.org/10.1016/j.apsusc.2017.09.190]

-
Chen, M., Qin, X. and Zeng, G., 2016, Single-walled Carbon Nanotube Release Affects the Microbial Enzyme-catalyzed Oxidation Processes of Organic Pollutants and Lignin Model Compounds in Nature, Chemosphere, Vol. 163, pp. 217~226.
[https://doi.org/10.1016/j.chemosphere.2016.08.031]

-
Liu, J., Xie, Y., Peng, C., Yu, G. and Zhou, J., 2017, Molecular Understanding of Laccase Adsorption on Charged Self-assembled Monolayers, The Journal of Physical Chemistry B, Vol. 121, No. 47, pp. 10610~10617.
[https://doi.org/10.1021/acs.jpcb.7b08738]

-
Yang, S., Liu, J., Quan, X. and Zhou, J., 2018, Bilirubin Oxidase Adsorption onto Charged Self-assembled Monolayers: Insights from Multiscale Simulations. Langmuir, Vol. 34, No. 33, pp. 9818~9828.
[https://doi.org/10.1021/acs.langmuir.8b01974]

-
Piontek, K., Antorini, M. and Choinowski, T., 2002, Crystal Structure of a Laccase from the Fungus Trametes Versicolor at 1.90-Å Resolution Containing a Full Complement of Coppers, Journal of Biological Chemistry, Vol. 277, No. 40, pp. 37663~37669.
[https://doi.org/10.1074/jbc.M204571200]

-
Pardo, I. and Camarero, S., 2015, Laccase Engineering by Rational and Evolutionary Design, Cellular and Molecular Life Sciences, Vol. 72, No. 5, pp. 897~910.
[https://doi.org/10.1007/s00018-014-1824-8]

-
Hong, G., Ivnitski, D. M., Johnson, G. R., Atanassov, P. and Pachter, R., 2011, Design Parameters for Tuning the Type 1 Cu Multicopper Oxidase Redox Potential: Insight from a Combination of First Principles and Empirical Molecular Dynamics Simulations, Journal of the American Chemical Society, Vol. 133, No. 13, pp. 4802~4809.
[https://doi.org/10.1021/ja105586q]

-
Sakurai, T. and Kataoka, K., 2007, Structure and Function of Type I Copper in Multicopper Oxidases, Cellular and Molecular Life Sciences, Vol. 64, No. 19, pp. 2642~2656.
[https://doi.org/10.1007/s00018-007-7183-y]

-
Lucas, M. F., Monza, E., Jørgensen, L. J., Ernst, H. A., Piontek, K., Bjerrum, M. J. et al., 2017, Simulating Substrate Recognition and Oxidation in Laccases: from Description to Design. Journal of Chemical Theory and Computation, Vol. 13, No. 3, pp. 1462~1467.
[https://doi.org/10.1021/acs.jctc.6b01158]

-
Santiago, G., De Salas, F., Lucas, M. F., Monza, E., Acebes, S., Martinez, A. T. et al., 2016, Computer-aided Laccase Engineering: Toward Biological Oxidation of Arylamines, ACS Catalysis, Vol. 6, No. 8, pp. 5415~5423.
[https://doi.org/10.1021/acscatal.6b01460]

-
Götze, J. P. and Bühl, M., 2016, Laccase Redox Potentials: pH Dependence and Mutants, a QM/MM Study, Journal of Physical Chemistry B, Vol. 120, No. 35, pp. 9265~9276.
[https://doi.org/10.1021/acs.jpcb.6b04978]

-
Monza, E., Lucas, M. F., Camarero, S., Alejaldre, L. C., Martínez, A. T. and Guallar, V., 2015, Insights into Laccase Engineering from Molecular Simulations: Toward a Binding-focused Strategy, Journal of Physical Chemistry Letters, Vol. 6, No. 8, pp. 1447~1453.
[https://doi.org/10.1021/acs.jpclett.5b00225]

-
Abraham, M. J., Murtola, T., Schulz, R., Páll, S., Smith, J. C., Hess, B. and Lindahl, E., 2015, GROMACS: High Performance Molecular Simulations through Multi-level Parallelism from Laptops to Supercomputers, SoftwareX, Vol. 1, pp. 19~25.
[https://doi.org/10.1016/j.softx.2015.06.001]

-
Xu, F., Palmer, A. E., Yaver, D. S., Berka, R. M., Gambetta, G. A., Brown, S. H. and Solomon, E. I., 1999, Targeted Mutations in a Trametes Villosa Laccase: Axial Perturbations of the T1 Copper. Journal of Biological Chemistry, Vol. 274, No. 18, pp. 12372~12375.
[https://doi.org/10.1074/jbc.274.18.12372]

-
Humphrey, W., Dalke, A. and Schulten, K., 1996, VMD: Visual Molecular Dynamics, Journal of Molecular Graphics, Vol. 14, No. 1, pp. 33~38.
[https://doi.org/10.1016/0263-7855(96)00018-5]

-
Kim, Y., Lee, M., Baek, I., Yoon, T. and Na, S., 2018, Mechanically Inferior Constituents in Spider Silk Result in Mechanically Superior Fibres by Adaptation to Harsh Hydration Conditions: A Molecular Dynamics Study, Journal of the Royal Society Interface, Vol. 15, No. 144, 20180305.
[https://doi.org/10.1098/rsif.2018.0305]

-
Choi, H., Lee, M., Park, H. S. and Na, S., 2016, The Effect of Structural Heterogeneity on the Conformation and Stability of Aβ–tau Mixtures, RSC Advances, Vol. 6, No. 57, pp. 52236~52247.
[https://doi.org/10.1039/C6RA09467H]

-
Choi, H., Chang, H. J., Lee, M. and Na, S., 2017, Characterizing Structural Stability of Amyloid Motif Fibrils Mediated by Water Molecules, ChemPhysChem, Vol. 18, No. 7, pp. 817~827.
[https://doi.org/10.1002/cphc.201601327]

-
Schmid, N., Eichenberger, A. P., Choutko, A., Riniker, S., Winger, M., Mark, A. E. and van Gunsteren, W. F., 2011, Definition and Testing of the GROMOS Force-field Versions 54A7 and 54B7, European Biophysics Journal, Vol. 40, No. 7, pp. 843~856.
[https://doi.org/10.1007/s00249-011-0700-9]

-
Miller III, B. R., McGee Jr, T. D., Swails, J. M., Homeyer, N., Gohlke, H. and Roitberg, A. E., 2012, MMPBSA. py: An Efficient Program for End-state Free Energy Calculations, Journal of Chemical Theory and Computation, Vol. 8, No. 9, pp. 3314~3321.
[https://doi.org/10.1021/ct300418h]

-
Brown, S. P. and Muchmore, S. W., 2006, High-throughput Calculation of Protein−ligand Binding Affinities: Modification and Adaptation of the MM-PBSA Protocol to Enterprise Grid Computing, Journal of Chemical Information and Modeling, Vol. 46, No. 3, pp. 999~1005.
[https://doi.org/10.1021/ci050488t]

-
Wong, S., Amaro, R. E. and McCammon, J. A., 2009, MM-PBSA Captures Key Role of Intercalating Water Molecules at a Protein−protein Interface, Journal of Chemical Theory and Computation, Vol. 5, No. 2, pp. 422~429.
[https://doi.org/10.1021/ct8003707]

-
Kumari, R., Kumar, R., Open Source Drug Discovery Consortium and Lynn, A., 2014, g_mmpbsa: A GROMACS Tool for High-throughput MM-PBSA Calculations, Journal of Chemical Information and Modeling, Vol. 54, No. 7, pp. 1951~1962.
[https://doi.org/10.1021/ci500020m]

-
Hebie, S., Holade, Y., Maximova, K., Sentis, M., Delaporte, P. et al., 2015, Advanced Electrocatalysts on the Basis of Bare Au Nanomaterials for Biofuel Cell Applications, ACS Catalysis, Vol. 5, No. 11, pp. 6489~6496.
[https://doi.org/10.1021/acscatal.5b01478]

-
Miyake, T., Yoshino, S., Yamada, T., Hata, K. and Nishizawa, M., 2011, Self-regulating Enzyme− nanotube Ensemble Films and Their Application as Flexible Electrodes for Biofuel Cells, Journal of the American Chemical Society, Vol. 133, No. 13, pp. 5129~5134.
[https://doi.org/10.1021/ja111517e]

-
Bandyopadhyaya, R., Nativ-Roth, E., Regev, O. and Yerushalmi-Rozen, R., 2002, Stabilization of Individual Carbon Nanotubes in Aqueous Solutions, Nano Letters, Vol. 2, No. 1, pp. 25~28.
[https://doi.org/10.1021/nl010065f]

-
An, K. H., Kim, W. S., Park, Y. S., Moon, J. M., Bae, D. J. et al., 2001, Electrochemical Properties of High‐power Supercapacitors Using Single‐walled Carbon Nanotube Electrodes, Advanced Functional Materials, Vol. 11, No. 5, pp. 387~392.
[https://doi.org/10.1002/1616-3028(200110)11:5<387::AID-ADFM387>3.0.CO;2-G]

-
Laviron, E., 1979, General Expression of the Linear Potential Sweep Voltammogram in the Case of Diffusionless Electrochemical Systems. Journal of Electroanalytical Chemistry and Interfacial Electrochemistry, Vol. 101, No. 1, pp. 19~28.
[https://doi.org/10.1016/S0022-0728(79)80075-3]

-
Allen, P. L. and Hickling, A., 1957, Electrochemistry of Sulphur: Part 1. — Overpotential in the Discharge of the Sulphide Ion, Transactions of the Faraday Society, Vol. 53, pp. 1626~1635.
[https://doi.org/10.1039/TF9575301626]

- Bard, A. J. and Faulkner, L. R., 2001, Fundamentals and Applications, Electrochemical Methods, Vol. 2, No. 482, pp. 580~632.
-
Frasconi, M., Boer, H., Koivula, A. and Mazzei, F., 2010, Electrochemical Evaluation of Electron Transfer Kinetics of High and Low Redox Potential Laccases on Gold Electrode Surface, Electrochimica Acta, No. 56, No. 2, pp. 817~827.
[https://doi.org/10.1016/j.electacta.2010.09.056]

-
ElKaoutit, M., Naranjo-Rodriguez, I., Temsamani, K. R., Domínguez, M. and de Cisneros, J. L. H. H., 2008, Investigation of Biosensor Signal Bioamplification: Comparison of Direct Electrochemistry Phenomena of Individual Laccase, and Dual Laccase-tyrosinase Copper Enzymes, at a Sonogel-carbon Electrode, Talanta, Vol. 75, No. 5, pp. 1348~1355.
[https://doi.org/10.1016/j.talanta.2008.01.055]

-
Navaee, A. and Salimi, A., 2015, Graphene-supported Pyrene-functionalized Amino-carbon Nanotube: A Novel Hybrid Architecture of Laccase Immobilization as Effective Bioelectrocatalyst for Oxygen Reduction Reaction, Journal of Materials Chemistry A, Vol. 3, No. 14, pp. 7623~7630.
[https://doi.org/10.1039/C4TA06867J]

-
Vasilescu, I., Eremia, S. A., Kusko, M., Radoi, A., Vasile, E. and Radu, G. L., 2016, Molybdenum Disulphide and Graphene Quantum Dots as Electrode Modifiers for Laccase Biosensor, Biosensors and Bioelectronics, Vol. 75, pp. 232~237.
[https://doi.org/10.1016/j.bios.2015.08.051]

-
Christenson, A., Dimcheva, N., Ferapontova, E. E., Gorton, L., Ruzgas, T. et al., 2004, Direct Electron Transfer between Ligninolytic Redox Enzymes and Electrodes, Electroanalysis, Vol. 16, No. 13‐14, pp. 1074~1092.
[https://doi.org/10.1002/elan.200403004]

-
Trohalaki, S., Pachter, R., Luckarift, H. R. and Johnson, G. R., 2012, Immobilization of the Laccases from Trametes Versicolor and Streptomyces Coelicolor on Single‐wall Carbon Nanotube Electrodes: A Molecular Dynamics Study, Fuel Cells, Vol. 12, No. 4, pp. 656~664.
[https://doi.org/10.1002/fuce.201200043]

-
Pettersen, E. F., Goddard, T. D., Huang, C. C., Couch, G. S., Greenblatt, D. M., Meng, E. C. and Ferrin, T. E., 2004, UCSF Chimera: A Visualization System for Exploratory Research and Analysis, Journal of Computational Chemistry, Vol. 25, No. 13, pp. 1605~1612.
[https://doi.org/10.1002/jcc.20084]

-
Rose, G. D., Geselowitz, A. R., Lesser, G. J., Lee, R. H. and Zehfus, M. H., 1985, Hydrophobicity of Amino Acid Residues in Globular Proteins, Science, Vol. 229, No. 4716, pp. 834~838.
[https://doi.org/10.1126/science.4023714]

-
Hush, N. S., 1985, Distance Dependence of Electron Transfer Rates, Coordination Chemistry Reviews, Vol. 64, pp. 135~157.
[https://doi.org/10.1016/0010-8545(85)80047-3]

-
Degani, Y. and Heller, A., 1987, Direct Electrical Communication between Chemically Modified Enzymes and Metal Electrodes. I. Electron Transfer from Glucose Oxidase to Metal Electrodes Via Electron Relays, Bound Covalently to the Enzyme, Journal of Physical Chemistry, Vol. 91, No. 6, pp. 1285~1289.
[https://doi.org/10.1021/j100290a001]

-
Degani, Y. and Heller, A., 1988, Direct Electrical Communication between Chemically Modified Enzymes and Metal electrodes. 2. Methods for Bonding Electron-transfer Relays to Glucose Oxidase and D-amino-acid Oxidase, Journal of the American Chemical Society, Vol. 110, No. 8, pp. 2615~2620.
[https://doi.org/10.1021/ja00216a040]

-
Martins, M. V., Pereira, A. R., Luz, R. A., Iost, R. M. and Crespilho, F. N., 2014, Evidence of Short-range Electron Transfer of a Redox Enzyme on Graphene Oxide Electrodes, Physical Chemistry Chemical Physics, Vol. 16, pp. 17426~17436.
[https://doi.org/10.1039/C4CP00452C]

- Kwon, J., Kim, J, I., Baek, I. and Na, S., 2015, Applying Mechanical Vibration and Coarse-grained Methods to Analyze Mechanical Properties of Actin Filaments, Proceedings of the KSNVE Annual Spring Conference, p. 187.
- Na, S.-S., Kim, Y.-J., Choi, H.-S., Chang H.-S., Yoon, T.-Y. and Park, W.-B., 2020, Mechanical Property-enhancing Mechanism of Silk Fiber through Synergy of Low Energy Accumulation and Self-regeneration, Proceedings of the KSNVE Annual Autumn Conference, p. 262.

Taeyoung Yoon is from Republic of Korea. After graduating from Korea University, he obtained combined master’s and doctorate course from Korea University in school of engineering science and mechanics. His research interests are in computational mechanics, molecular dynamics and biofuel cells with electrochemical experiments.

Sungsoo Na is a professor in the Department of Engineering Science and Mechanics at Korea University. He received B.S. and M.S. in engineering science and mechanics at Korea University and received Ph.D. in engineering science and mechanics at Virginia Polytechnic Institue & State University. His principle research interests include detection of toxic nanomaterial and protein dynamics.